基因组注释软件TriAnnot安装配置
基因组注释(Genome annotation) 是利用生物信息学方法和工具,对基因组所有基因的生物学功能进行高通量注释,是当前功能基因组学研究的一个热点。
基因组注释的研究内容包括基因识别和基因功能注释两个方面。
TriAnnot是著名的基因组注释软件之一。
软件主页:https://sourcesup.renater.fr/wiki/triannot/index
论文链接:http://europepmc.org/abstract/MED/22645565
本次安装配置的测试环境是无root权限的ubuntu16.04服务器,只预装了最基本的编译环境。
以下命令默认安装目录为/home/zhu/Documents/TriAnnot/
官方表示发邮件可以获取最新源码:
You can request the latest TriAnnot package by sending an email to triannot-support@clermont.inra.fr
本次使用的版本为triannotV5.2p02
下面进入安装:
1.下载软件、依赖、数据库
根据不同的任务需求,需要不同的软件和数据库,依次下载放到目标路径。
主要包括三个文件夹
/home/zhu/Documents/TriAnnot/sourcecode_triannotV5.2p02
/home/zhu/Documents/TriAnnot/TriAnnot_software
/home/zhu/Documents/TriAnnot/TriAnnot_databank
添加主程序环境变量
vi ~/.bashrc
在其中加入
export PATH=/home/zhu/Documents/TriAnnot/sourcecode_triannotV5.2p02/bin:$PATH
export PYTHONPATH=/home/zhu/Documents/TriAnnot/sourcecode_triannotV5.2p02/pythonlib:$PYTHONPATH
export PERL5LIB=/home/zhu/Documents/TriAnnot/sourcecode_triannotV5.2p02/perllib/:$PERL5LIB
使环境变量生效
source ~/.bashrc
python /home/zhu/Documents/TriAnnot/sourcecode_triannotV5.2p02/bin/TriAnnotPipeline.py -w Check run --check -s A20.tfa -t wheat_step161213-long.xml --type nucleic
需要根据测试中报告的错误修改各个软件和数据库的路径。(每次测试后需要删除Check文件夹才能进行下一次测试)
2.编译软件
如果服务器无root权限有网络,推荐软件安装神器linuxbrew
需要安装git、curl
git clone https://github.com/Linuxbrew/brew.git ~/.linuxbrew cd ~/.linuxbrew
修改环境变量
vi ~/.bashrc
在其中加入
export PATH=$HOME/.linuxbrew/bin:$PATH
export MANPATH="$(brew --prefix)/share/man:$MANPATH"
export INFOPATH="$(brew --prefix)/share/info:$INFOPATH"
export CPLUS_INCLUDE_PATH=$HOME/.linuxbrew/include/:$CPLUS_INCLUDE_PATH
export C_INCLUDE_PATH=$HOME/.linuxbrew/include/:$C_INCLUDE_PATH
export LD_LIBRARY_PATH=$HOME/.linuxbrew/lib/:$LD_LIBRARY_PATH
export LIBRARY_PATH=$HOME/.linuxbrew/lib/:$LIBRARY_PATH
此处需要注意所有指向linuxbrew的环境变量最好放在最后,否则可能在TriAnnot运行时优先找到linuxbrew目录导致运行失败。
使环境变量生效
source ~/.bashrc
git -C "$(brew --repo homebrew/core)" fetch --unshallow
然后brew install XXXXX 即可安装各种软件
因为下面每个软件包都有很多依赖库,自己编译安装极其复杂,在硬盘空间有较多空闲的情况下,推荐使用brew install xxxxxx先安装一次,几乎都可以自动装好所有的依赖库,大大减少工作时间。
RepeatMasker配置
brew install Repeatmasker
首先添加环境变量
export PATH=/TriAnnot_software/RepeatMasker/:$PATH
然后进入软件目录配置
cd /home/zhu/Documents/TriAnnot/TriAnnot_software/RepeatMasker ./configure
指定perl目录(一般默认即可)
指定RepeatMasker目录(/TriAnnot_software/RepeatMasker)
指定trf目录(/TriAnnot_software/trf404.linux)
然后配置工具
先配置1.crossmatch
路径为/TriAnnot_software/phrap_cross_match_swat/
确定后输入y设置为默认
再配置4.hmmer-3
路径为/TriAnnot_software/hmmer-3.1b2-linux-intel-x86_64/binaries/
确定后输入n不设置为默认
最后输入5.done,即为配置完成
接下来安装perl依赖库
使用cpan可以方便的安装,没有cpan的机器就只能自行百度了。
需要的库:
cpan Text::Soundex
cpan Capture::Tiny
cpan Switch
cpan Bio::SeqIO
安装完成后,命令行输入RepeatMasker可以显示帮助菜单即为安装成功。
rnammer配置
brew install rnammer vi /home/zhu/Documents/TriAnnot/TriAnnot_software/rnammer-1.2.src/rnammer
修改35行:my $INSTALL_PATH=/home/zhu/Documents/TriAnnot/TriAnnot_software/rnammer-1.2.src
51行:$HMMSEARCH_BINARY = "/home/zhu/Documents/TriAnnot/TriAnnot_software/hmmer-3.1b2-linux-intel-x86_64/binaries/hmmsearch";
54行:$HMMSEARCH_BINARY = "/home/zhu/Documents/TriAnnot/TriAnnot_software/hmmer-3.1b2-linux-intel-x86_64/binaries/hmmsearch";
getorf配置
getrof是EMBOSS软件里的子模块。
brew install emboss
然后进入TriAnnot_software文件夹下编译安装。
缺少libmysqlclient.so、libssl.so等一大堆动态链接库,因此决定直接替换为brew安装好的版本。
mv /home/zhu/Documents/TriAnnot/TriAnnot_software/EMBOSS-6.6.0 /home/zhu/Documents/TriAnnot/TriAnnot_software/EMBOSS-6.6.0_old cp -r ~/.linuxbrew/Cellar/emboss/6.6.0/ /home/zhu/Documents/TriAnnot/TriAnnot_software/EMBOSS-6.6.0/
添加环境变量
export PATH=/home/zhu/Documents/TriAnnot/TriAnnot_software/EMBOSS-6.6.0/bin/:$PATH
命令行输入which getorf 如果指向此路径即为成功
InterProScan5配置
本软件需要java环境
brew install jdk
添加环境变量
export $JAVA_HOME=$HOME/.linuxbrew/Cellar/jdk/1.8.0-144/:$JAVA_HOME
Blast配置
BestHit模块缺少Perl库 Bio::SearchIO::blastxml
(执行下句中出现无限的找不到apache的错误提示,可以使用ctrl+c退出)
cpan Bio::SearchIO::blastxml
错误在仿佛是在安装SOAP-Lite-1.22过程中出现
再次运行安装,报错
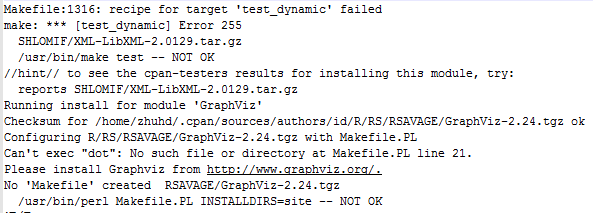
运行
brew install GraphViz cpan GraphViz
安装成功
继续安装cpan Bio::SearchIO::blastxml
自动安装依赖XML::LibXML时出错
cpan XML::LibXML
依然有错
然而到此处Blast的错误仿佛迷之解决了...
(以上均为后期可以运行TriAnnot主程序之后出现的错误解决方案,其余所有的C++软件均需要自行编译,编译过程没有记录)
tRNAscanSE配置
具体过程没有记录,看起来仿佛挺心酸的
最终解决方案是把配置文件中的路径改到了/home/zhu/.linuxbrew/bin/tRNAscan-SE
3.路径配置
主要需要修改的配置文件为:/home/zhu/Documents/TriAnnot/sourcecode_triannotV5.2p02/conf
分为两大部分
software和databank
databank基本都是通过defaultDbPath变量指定的,只需改第9行即可。
software需要一一修改,指定为/TriAnnot_software/文件夹下的对应软件
最后的config里Eugene和Augustus也同样需要修改
python /home/zhu/Documents/TriAnnot/sourcecode_triannotV5.2p02/bin/TriAnnotPipeline.py -w Check run --check -s A20.tfa -t wheat_step161213-long.xml --type nucleic
执行测试命令,如果出现以下输出(没有ERROR),则为配置成功。
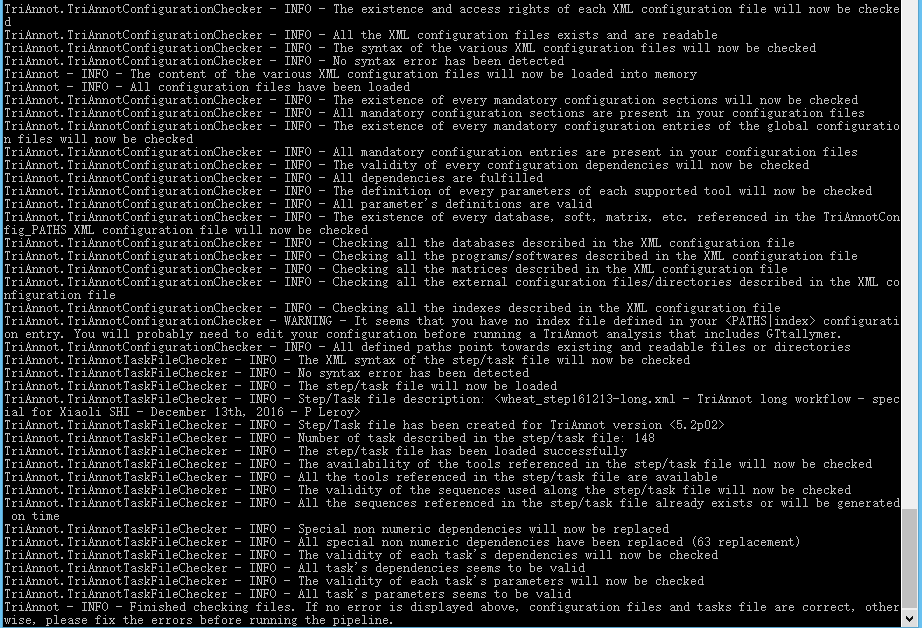
4.测试运行
运行命令为
/home/zhu/Documents/TriAnnot/sourcecode_triannotV5.2p02/bin/TriAnnotPipeline.py -w Anno_scaffold4605 --debug --logtofile run -s scaffold4605.fasta -t wheat_step161213-long.xml --type nucleic --clean loetcsp --mth-override 4 --minlength 50000 --maxlength 500000 --splitseq --overlap 50000 &> runscaffold4605.log
good luck~